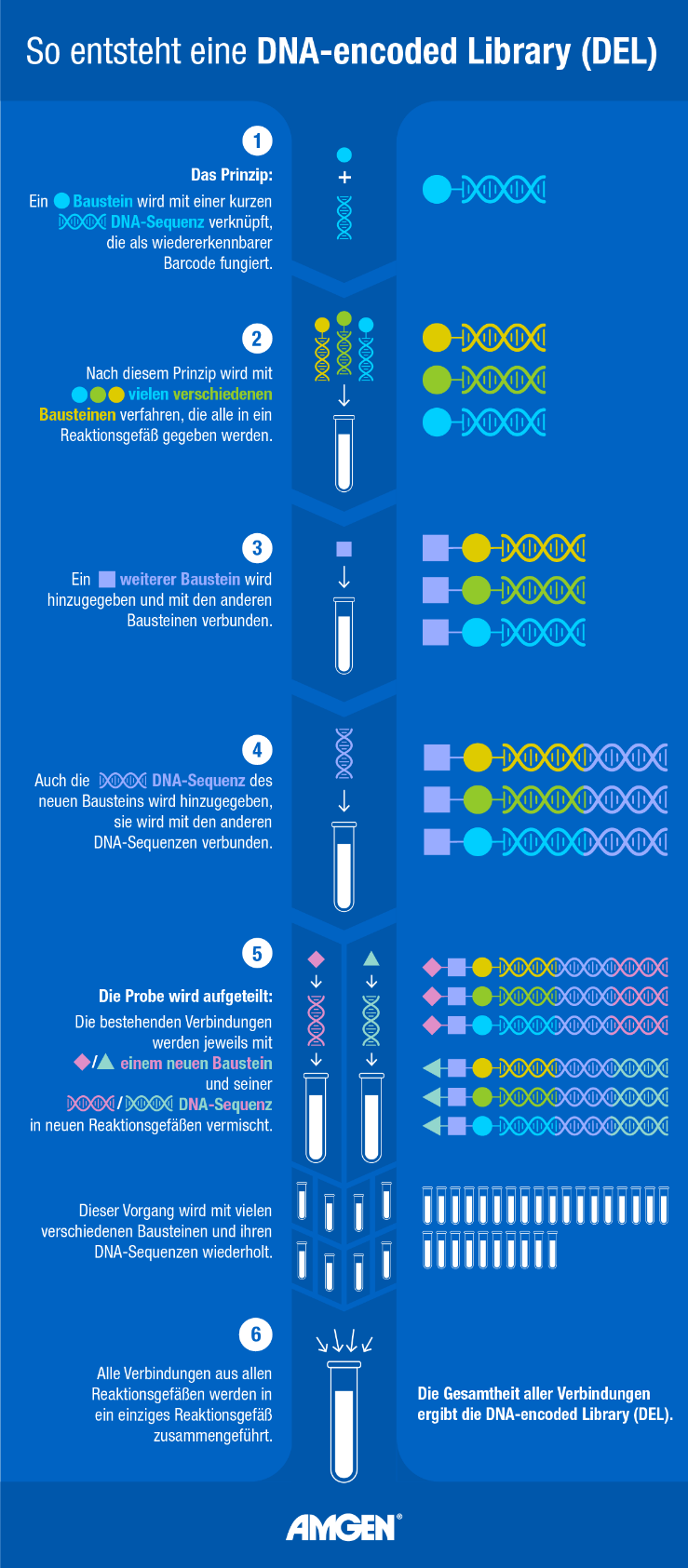
Amgen hat es sich zur Aufgabe gemacht, Arzneimittel für bisher unheilbare Erkrankungen zu entwickeln. Ein zentrales neues Werkzeug sind dabei DNA-kodierte Bibliotheken (engl. DNA-encoded Libraries, kurz DEL). Mit dieser Technologie können bis zu eine Million Mal mehr potenzielle Wirkstoffe gleichzeitig getestet werden als mit herkömmlichen Methoden1. So wird die Chance vervielfacht, einen erfolgversprechenden Wirkstoff zu finden und ein innovatives Arzneimittel zu entwickeln.

Warum DEL?
Jeder Mensch besitzt mehr als 30.000 Proteine, die verschiedene Aufgaben im Körper erfüllen. Ist die Funktion eines Proteins gestört, kann das zu einer Erkrankung führen. Viele dieser Fehlfunktionen sind aber schwer oder gar nicht therapierbar: Für etwa 80 Prozent der abweichenden Strukturen konnten Forscherinnen und Forscher bisher keinen Wirkstoff finden, der an sie bindet, die physiologische Funktion wieder herstellt bzw. die Fehlfunktion behebt und so einer möglichen Erkrankung Einhalt gebieten könnte. Solche therapeutisch nicht angreifbaren Proteine werden als „undruggable“ bezeichnet2.
Bei der Suche nach Wirkstoffen, die an solche Proteine binden können, ist die Nutzung einer DNA-encoded Library von Vorteil. Analysiert wird dabei eine bestimmte Art von Wirkstoffen, die sogenannten small molecules (dt. kleine Moleküle oder niedermolekulare Verbindungen). Sie bestehen aus verschiedenen Molekülen, auch als Bausteine bezeichnet, die aneinander binden. Während durch konventionelle Hochdurchsatz-Methoden (engl. High-throughput screening, kurz HTS) etwa 10.000 dieser unterschiedlichen Molekülverbindungen pro Woche getestet werden können3, ermöglicht eine DNA-encoded Library, Milliarden verschiedener Kandidaten gleichzeitig zu analysieren1. Das spart Zeit und Kosten, denn obwohl Hochdurchsatz-Methoden automatisiert sind, können sie mit der Effizienz einer DNA-encoded Library nicht mithalten. Wie ist das möglich?
Was ist die Besonderheit der DEL-Technologie?
Die Gesamtheit der zu untersuchenden Molekülverbindungen ergeben die Bibliothek (Library), aus der sich im besten Fall ein Wirkstoff identifizieren lässt. Die DEL-Technologie ermöglicht es, alle potenziellen Wirkstoffe, also die komplette Bibliothek, auf einmal und im selben Reaktionsgefäß zu testen. Bei der HTS-Methode hingegen werden die unterschiedlichen Verbindungen voneinander getrennt und die Wirkung jedes einzelnen small molecules auf das Zielprotein wird gesondert untersucht4; 5. Eine Million Verbindungen in Kombination mit 50 verschiedenen Zielproteinen zu testen, benötigt folglich mit herkömmlichen Methoden 50 Millionen Ansätze in 50 Millionen Reaktionsgefäßen. Mit der DEL-Technologie sind nur 50 Ansätze nötig.
Das Besondere der DEL-Technologie ist, dass jede zu testende Molekülverbindung mit einer einzigartigen DNA-Sequenz verknüpft ist. Diese Sequenz ist gleichzeitig der Barcode der Verbindung. Sie ermöglicht es, ein spezielles Molekül in einer Mischung aus Millionen verschiedener Moleküle sicher zu identifizieren. Daher müssen die zu testenden small molecules nicht auf verschiedene Reaktionsgefäße aufgeteilt werden, sondern können alle im selben Versuchsansatz analysiert werden.
Nach Zugabe des Zielproteins zur Molekülbibliothek werden diejenigen Verbindungen isoliert, die an das Zielprotein binden. Diese Verbindungen sind für die weitere Entwicklungsarbeit interessant. Im nächsten Schritt werden die Sequenzen der DNA-Codes dieser Verbindungen bestimmt. Das ist mit gängigen molekularbiologischen Methoden schnell und einfach möglich. Zielsicher lässt sich nun auf die small molecules schließen, die an das Protein gebunden haben. Diese Verbindungen sind potenzielle Kandidaten für die Entwicklung eines Arzneimittels4; 5.
Eine perfekte Technologie?
Auch die DEL-Technologie hat ihre Grenzen. Zwar können die Forscherinnen und Forscher durch den Barcode effektiv die small molecules identifizieren, die an das Protein binden. Doch welche Auswirkung diese Bindung auf das Zielprotein hat, bleibt dabei erst einmal offen. Blockt das small molecule die Funktion des Zielproteins? Steigert es seine Aktivität? Verhindert es, dass andere Proteine mit dem Zielprotein interagieren können? Antworten auf diese Fragen können nur Untersuchungen mit herkömmlichen Methoden liefern.
Potential für Amgen
Mithilfe der DEL-Technologie können unter anderem bispezifische Moleküle identifiziert werden. Sie bestehen aus zwei unterschiedlichen Verbindungen, die durch DEL aufgespürt und aneinander gekoppelt werden können. Solche Moleküle können gleichzeitig an zwei verschiedene Proteine binden, daher auch die Bezeichnung „bispezifisch“. Ein Beispiel für bispezifische small molecules sind PROTACs (engl. Proteolysis targeting chimera). Sie verbinden ein Zielprotein mit der Ubiquitin Ligase, einem körpereigenen Enzym des Proteinabbaus. Die Ligase markiert das Zielprotein für den Abbau durch das Proteasom, die „Müllabfuhr“ der Zellen. Bispezifische small molecules können also an die unterschiedlichsten Proteine binden und so zukünftig bei der Entwicklung von Therapeutika gegen eine Reihe von Erkrankungen angewendet werden6.
Jedes Protein im Körper interagiert von Natur aus mit einer Vielzahl anderer Proteine. Viele dieser Wechselbeziehungen und ihre Bedeutung für bestimmte Erkrankungen sind heute bekannt. Durch bispezifische Moleküle können die Interaktionen gezielt beeinflusst werden, um die Funktion gestörter Proteine zu kontrollieren. „Mit Hilfe der DEL-Technologie können wir therapeutisch sonst kaum beeinflussbare Zielproteine mit solchen Proteinen verbinden, die dadurch ihren natürlichen Effekt auf das Zielprotein ausüben können“, erklärt Dr. Stefan Kropff, Medizinischer Direktor der Amgen GmbH. So kann die Aktivität oder Struktur des krankmachenden Proteins verändert werden, ohne dass die small molecules selbst eine Wirkung entfalten müssen. Durch die DEL-Technologie wird es zukünftig einfacher sein, bispezifische Verbindungen zu entwickeln, um sie gegen verschiedene Erkrankungen einzusetzen
Hintergrundinformationen
Referenzen:
1. Wrenn S. J. et al. Synthetic ligands discovered by in vitro selection. J Am Chem Soc. 2007; 129 (43): 13137–13143. https://doi.org/10.1021/ja073993a.
2. Crews C. M. Targeting the undruggable proteome: the small molecules of my dreams. Chem Biol. 2010; 17 (6): 551–555. https://doi.org/10.1016/j.chembiol.2010.05.011.
3. Macarron R., Banks M., Bojanic D. et al. Impact of high-throughput screening in biomedical research. Nat Rev Drug Discov 2011; 10: 188–195. https://doi.org/10.1038/nrd3368.
4. Franzini R. M., Neri D., Scheuermann J. DNA-encoded chemical libraries: advancing beyond conventional small-molecule libraries. Acc. Chem. Res. 2014; 47, 1247−1255. https://doi.org/10.1021/ar400284t.
5. Mannocci L., Leimbacher M., Wichert M., Scheuermann J., Neri D. 20 years of DNA-encoded chemical libraries. Chem. Commun. 2011; 47, 12747–12753. https://doi.org/10.1039/c1cc15634a.
6. Neklesa T. K., Winkler J. D., Crews C. M. Targeted protein degradation by PROTACs. Pharmacology & therapeutics 2017; 174: 138-144. http://dx.doi.org/10.1016/j.pharmthera.2017.02.027.
7. vfa Biotech Report 2020
8. Krall N., Scheuermann J., Neri D. Small targeted cytotoxics: current state and promises from DNA‐encoded chemical libraries. Angew. Chem. Int. Ed. 2013, 52, 1384 – 1402. https://doi.org/10.1002/anie.201204631.
15.04.2021